Muchas personas poseen mutaciones que causan enfermedades, sin embargo, nunca las desarrollan. Investigadores del Centro de Regulación Genómica (CRG) han estudiado cómo las variaciones en la expresión de genes pueden predecir si un individuo desarrollará o no una enfermedad. los expertos concluyen que la información del genoma no será suficiente para predecir qué le ocurrirá a cada persona.
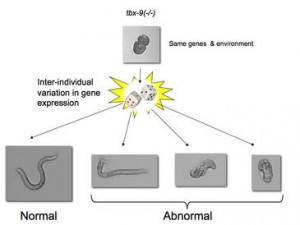
La misma mutación puede afectar de modo diferente a cada individuo de C. elegans . Los dados representan el componente estocástico en la expresión de genes. Imagen: CRG
La gran mayoría de mutaciones que causan enfermedades (esquizofrenia o cáncer de mama, por ejemplo) tienen efectos diferentes según la persona. Un individuo que posee determinadas mutaciones puede desarrollar la enfermedad, mientras que otro con las mismas mutaciones puede no hacerlo.
Este hecho, es evidente si se compara a dos gemelos monocigóticos, cuyos genomas son idénticos. Pero, ¿porqué la misma mutación tiene efectos distintos en cada persona?
“Estudiando organismos muy simples, como bacterias, sabemos que el grado de activación o represión de un gen varía mucho entre individuos, incluso en ausencia de variación genética y ambiental” explica Alejandro Burga, investigador del CRG y co-autor del estudio que se publica en la revista Nature.
Una investigación, en la que han participado científicos del CRG sugiere que aunque lleguemos a conocer todos los genes importantes para una enfermedad en particular, quizá no podremos predecir que le pasará a cada persona contando únicamente con la información de su genoma.
Para poder desarrollar una medicina personalizada y predictiva será también necesario considerar el grado de variación en que los genes son activados y reprimidos en cada persona.
Dos células nunca son exactamente iguales y en ocasiones, estas diferencias tienen un componente azarso o estocástico. Los resultados del estudio muestran que este tipo de variaciónes “puede influir de manera importante en el fenotipo de animales y que su medición ayuda a predecir con mucha mayor fiabilidad la probabilidad de presentar un fenotipo anormal, como por ejemplo, el desarrollo de una enfermedad” añade Burga.
Desde principios del siglo XX se ha estudiado el papel que tienen las mutaciones y el ambiente (hábitos de consumo, estilo de vida, etc.) en el desarrollo de enfermedades. Aunque Burga apunta que “las diferencias genéticas y el ambiente no son suficientes”.
Dado que la composición genética y el ambiente son insuficientes para saber si una mutación afectara o no a un individuo, los investigadores desarrollaron una metodología para medir pequeñas diferencias en la expresión de genes in vivo.
“El reto no fue sólo el desarrollo de un método para medir estas pequeñas diferencias entre individuos, sino el poder predecir que genes son relevantes para cada mutación”, explica Ben Lehner, coordinador del estudio e investigador en la unidad de Biología de Sistemas del European Molecular Biology Laboratory del CRG.
Los autores del trabajo realizaron su estudio utilizando el gusano microscópico Caenorhabditis elegans, uno de los organismos modelos más usados en biología debido a su simplicidad.
La importancia de este organismo, que fue el primer animal en tener su genoma totalmente secuenciado, se demuestra en el hecho de que en los últimos años, el premio Nobel ha sido otorgado en tres ocasiones a investigadores que lo usaron en sus estudios.
“Tanto en gusanos como en humanos, los genes cooperan y se ayudan mutuamente para realizar funciones dentro de la célula. Unos pocos genes son muy ‘generosos’ y ayudan a cientos de otros en muchos procesos distintos, mientras que otros solo ayudan a unos pocos genes a desarrollar tareas más específicas. La clave para realizar mejores predicciones radica en medir las variaciones en la expresión de ambos tipos de genes”, explican los autores.
La investigación se realizó con fondos del European Research Council (ERC), Institució Catalana de recerca i Estudis Avanzats (ICREA) y el Ministerio de Ciencia e Innovación.
Referencia bibliográfica:
Burga, A; Casanueva, O; Lehner, B. “Predicting mutation outcome from early stochastic variation in genetic interaction partners”. Nature, 8th of December,. doi:10.1038/nature10665, 2011
CRG / SINC






