En el genoma, los genes que codifican por proteínas similares y que se regulan conjuntamente suelen encontrarse cerca. Pero también hay genes que no tienen esta particularidad y, aun así, se mantienen juntos en el genoma. Por qué se separan, o no, algunos genes, ligados ancestralmente en el genoma, es una incógnita que ahora tiene una nueva respuesta científica, según se desprende de un artículo publicado en la revista Genome Research.
El trabajo investigador, centrado en el ámbito de la arquitectura genómica, se basa en el estudio del gen Sosondowah.
Un estudio de la Universidad de Barelona demuestra, por primera vez, cómo pueden separarse los genes a lo largo de los linajes evolutivos mediante el mecanismo de la retrotransposición, que es la síntesis de ADN a partir de ARN a través de la transcriptasa inversa.
Firman el nuevo trabajo los investigadores Jordi Garcia-FernÁ ndez, Ignacio Maeso y Manuel Irimia, del Departamento de Genética y del Instituto de Biomedicina de la Universidad de Barcelona (IBUB), científicos de los equipos encabezados por José Luis Gómez-Skarmeta, del Centro Andaluz de Biología del Desarrollo, CSIC, y por Sonsoles Campuzano, del Centro de Biología Molecular Severo Ochoa, CSIC, y expertos de la Universidad Nacional de Singapur.
Transcriptasa inversa: del ARN al ADN
El genoma humano contiene muchas retrosecuencias, es decir, segmentos que son el resultado de la transcripción inversa del ARN y la integración del nuevo ADN en nuevos puntos del genoma. La transcriptasa inversa, una enzima que también participa en la síntesis y el mantenimiento de los telómeros (extremos de los cromosomas), es la responsable de este particular mecanismo molecular.
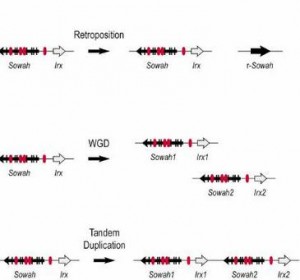
Como primicia, el nuevo trabajo describe de qué manera la retrotransposición también ha permitido desligar genes que estaban próximos desde hace millones de años en la escala evolutiva. «Hasta ahora, solo se conocían dos mecanismos para separar genes: la duplicación completa del genoma y la pérdida de una copia, y la duplicación en tándem y pérdida de una de las copias, unos procesos moleculares que han tenido lugar en distintos momentos de la evolución biológica. En el nuevo artículo, describimos por primera vez una nueva vía de separación entre genes», explica el catedrático Jordi Garcia-FernÁ ndez, director del equipo del Reg-Volution Lab de la UB.
Tras la pista del gen Sosondowah
El trabajo del Genome Research, centrado en el ámbito de la arquitectura genómica, se basa en el estudio del gen Sosondowah, que a escala topográfica es adyacente a los genes Iroquois, un complejo génico implicado en la neurogénesis del sistema nervioso en la Drosophilay en vertebrados. “Los genes Iroquois —apunta Garcia-FernÁ ndez— están flanqueados por extensas áreas que son desiertos génicos, es decir, zonas del genoma muy pobres en genes y con tendencia a duplicarse. En el genoma de los vertebrados, en concreto, hay más de 3.000 regiones reguladoras ultraconservadas, y algunas de estas zonas se ubican dentro del complejo Iroquois“.
Según los autores, para la mayoría de los metazoos estudiados, el gen Sosondowah (Sowah)estaría ligado a los Iroquois,porque Sowah contiene, en sus intrones (regiones no codificadoras), una serie de regiones reguladoras clave para los genes Iroquois. En los vertebrados, en cambio, Sosondowah se habría escapado mediante mecanismos de retrotransposición, en un escenario evolutivo descrito ahora como primicia, y caracterizado por la conservación de los intrones del gen Sosondowah y la pérdida de los exones (regiones codificadoras) a lo largo de la evolución biológica.
Referencia bibliográfica:
Ignacio Maeso, Manuel Irimia, Juan J Tena, Esther González-Pérez, David Tran, Vydianathan Ravi, Byrappa Venkatesh, Sonsoles Campuzano, José Luis Gómez-Skarmeta, Jordi Garcia-FernÁ ndez. “An ancient genomic regulatory block conserved across bilaterians and its dismantling in tetrapods by retrogene replacement”. Genome Res. Advance January 10, 2012, doi: 10.1101/gr.132233.111
UB / SINC






